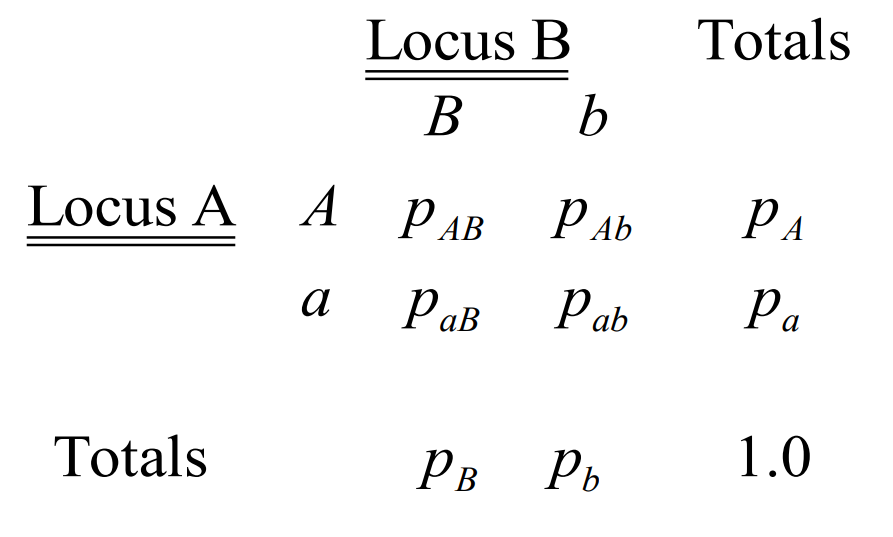
先用一个简答的例子,来理解linkage disequilibrium,即LD, “chromosomes are mosaics”,这句话如何理解? 当一段序列最初始的状态,呈现在眼前的时候,我们称其为“ancestral state”,即祖先状态, 就像人会经历各种各样的事情被改变,DNA序列也是一样的, 上述这些因素都是会造成现如今的DNA呈现在我们眼前如此多样的原因。 来看张图理解, 引入D统计量, Note:D存在多种变式,比如、 假设在某条染色体上存在两个相邻locus,第一个locus的gene有A和a,第二个locus的gene有B和b,那么它们在理想群体下的frequency是多少? 如果locus之间是存在linkage disequilibrium效应的呢?则会出现目标gamete type的期望频率和实际频率的偏离,用来衡量。以为例,
针对每种gamete type frequency的偏离,其计算表达式如下,
Note:如何理解? 默认情况下,将A、B看作是common allele,而a、b看作是rare allele即可反映上述关系。 该系数没有消除量纲,因此取值范围没有被限定。 为了更好的理解,连锁不平衡效应在代际变化下的变化趋势,就需要引入一个另外一个例子。 首先注意阐明一个点,由于现在是计算2个locus,为了表示代际之间的gene frequency、genotype frequency,需要引入另一个中间变量 —— gamete frequency。 为什么要引入? 举个例子,以携带AABB这个genotype的个体为例,该个体产生的gamete只存在AB这种类型, 那如果是携带AaBb这个genotype的个体呢?如果不提前表明配子类型如何,该如何知道,究竟是Ab还是AB呢? 因此就需要将AaBb这种情况给“phasing”开,即用AB/ab和Ab/aB这两种形式来表示。 开始推导。 假设在当前世代t,群体中能够产生或者说AB gamete占总gamete数的比例为,下一代,AB gamete的占比,是多少? 同时引入r,即recombination rate,表示配子之间的gene是可以互换的,就比如AB/ab原本只能够产生AB和ab这两种配子,但由于recombination,现在也可以产生Ab、aB另外两种配子,而重组发生的事件,我们定义为r。 那么下一代的即为,
变式,可以得到如下的式子,
而在t个世代之后的关系式,则为,
而随着t不断增大,等式右边趋近于0,即表示实际gamete frequency和期望frequency之间的差异消失了,LD不见了! 理解了这个,就理解了haplotype frequency如何变化。 从上述式子,再反推回来,我们可以得到如下的式子,
上述的式子非常好理解, 同时,在linkage equilibrium的情况下,使用gamete frequency可以帮助我们节省很多的计算量, 比如现存在20个locus,每一个locus存在2个allele,那么就可以组成3个genotype(不区分Aa和aA), 如果直接保留genotype的计算结果,那我们就需要从个genotype中计算最终的gene frequency,但是如果群体满足linkage equilibrium,我们则可以直接使用gamete type来计算gamete frequency,。 懒了,直接上图。 ,实际上是从gamete frequency计算genotype frequency再返回gamete frequency的做法 联系这张图,就可以一下子理解 而最终就可以得到,如下这样的一个关系式,
上述的式子反映了这样的一个生物学问题:群体中的LD主要由genotype为AB/ab、Ab/aB这两者之间的frequency差异造成,因此当达到linkage equilibrium时,它们之间的差异消失了,不同的gene之间的linkage也就不见了。 D'_{AB}\left\{
\begin{array}{**lr**}
\frac{D_{AB}}{min(p_{A}p_{B},p_{a}p_{b})}, D_{AB}<0 \\
\frac{D_{AB}}{min(p_{A}p_{b},p_{a}p_{B})}, D_{AB}>0
\end{array}
\right.
取值范围: 1,代表loci之间存在完全的连锁关系(e.g. 如果AB为初始gamete,且后续也不存在recombination,将它们之间的连锁关系给它们,那么在当前的群体中,只要出现了A,也就能确定B,即“perfect LD, it means the observation at one marker provides complete information about the other”) 0,代表linkage equilibrium [1] Felsenstein, J., 2005. Theoretical evolutionary genetics joseph felsenstein. University of Washington, Seattle. [2] Xu, S. and Jin, W., 2012. Population Genetics in the Genomic Era. Edited by M. Carmen Fusté, p.137. [3] Biostatistics 666, Abecasis Lab [4] Slatkin, M., 2008. Linkage disequilibrium—understanding the evolutionary past and mapping the medical future. Nature Reviews Genetics, 9(6), pp.477-485.
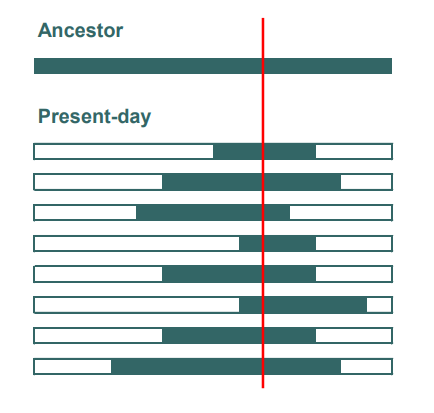
如何衡量Linkage Disequilibrium?
Linkage Disequilibrium会消失吗?| recombination rate的引入
gamete frequency怎么变化?
从LD的角度来理解gamete frequency变化
从gamete frequency本身来理解gameter frequency的变化


其他
参考资料
Hi,这里是有朴的第二大脑。
很高兴与你相遇
很高兴与你相遇